Genetyczny odcisk palca
Technika odkryta przez Aleca Jeffreysa w 1984 na Uniwersytecie Leicester pozwala identyfikować pewne wzory DNA w loci (l. mnoga od „locus”, rodzaj „pojemników” na konkretne sekwencje kodu), które są unikalne dla każdego człowieka za wyjątkiem bliźniąt jednojajowych. Każdy taki wzór to powtarzająca się część DNA (znana jako „zmienna liczba tandemowych powtórzeń” tzn. VNTR), której wielkość zależy od ilości powtórzeń. W granicach danego locus liczba takich powtórzeń jest różna u różnych osobników.
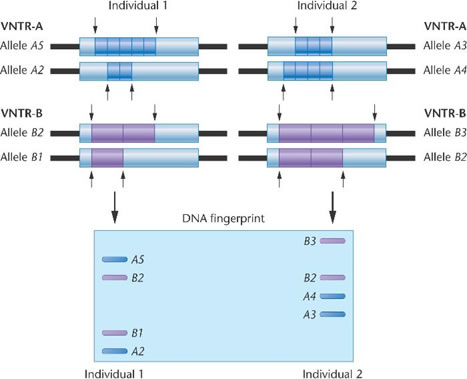
Rys 3.2 Przykład analizy VNTR
Źródło: http://bio3400.nicerweb.com/Locked/media/ch22/22_27-VNTR.jpg
W rezultacie proces ten bazuje na analizie RFLP (polimorfizm długości fragmentów restrykcyjnych), którą to techniką wykrywa się różnice pomiędzy rozmiarami fragmentów DNA pociętych restryktyzami.
Procedura utworzenia genetycznego odcisku palca składa się z następujących kroków:
- Izolacja DNA – konieczne jest uzyskanie próbki DNA. Należy w szczególności uważać na możliwość jej zanieczyszczenia.
- Cięcie, mierzenie oraz sortowanie – przy pomocy specjalnych enzymów zwanych restryktyzami próbka DNA zostaje pocięta w konkretnych miejscach, w związku z czym powtarzalne sekcje kodu, o których była wcześniej mowa, stają się widoczne. Następnie zostają one odseparowane oraz posortowane przy pomocy procesu elektroforezy żelazowej (istotą tej techniki jest rozdzielnie poprzez wymuszenie wędrówki cząsteczek w polu elektrycznym).
- Przeniesienie – uzyskany w ten sposób wynik jest nanoszony na odpowiednią błonę nylonową (tzw. Bloting DNA – jest to transfer DNA na błonę/filtr najczęściej w celu hybrydyzacji z próbką znakowanego DNA).
- Sondowanie – do otrzymanego związku dodajemy radioaktywne lub kolorujące cząsteczki znakującew celu uzyskania genetycznego odcisku palca.
- Faza końcowa – ostateczny wynik otrzymujemy przy pomocy ok. 5 do 10 takich cząsteczek – przypomina on pod pewnymi względami kod kreskowy.
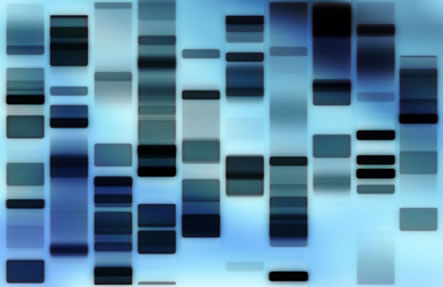
Rys 3.3 Genetyczny odcisk palca
Źródło: http://www.nuffieldbioethics.org/bioinformation